CH CRL (R4)
0.9.0 - Draft
CH CRL (R4)
0.9.0 - Draft
This page is part of the CH CRL (R4) (v0.9.0: STU Draft) based on FHIR R4. . For a full list of available versions, see the Directory of published versions 
| Defining URL: | http://fhir.ch/ig/ch-crl/StructureDefinition/ch-crl-cancerreport |
| Version: | 0.9.0 |
| Name: | CHCRLCancerReport |
| Title: | CH CRL Cancer Report Logical Model |
| Status: | Draft as of 2022-02-04 02:14:21+0100 |
| Definition: | Logical model of the cancer report |
| Publisher: | Bundesamt für Gesundheit BAG |
| Copyright: | CC-BY-SA-4.0 |
| Source Resource: | XML / JSON / Turtle |
The official URL for this profile is:
http://fhir.ch/ig/ch-crl/StructureDefinition/ch-crl-cancerreport
Description of Profiles, Differentials, Snapshots and how the different presentations work.
This structure is derived from Element
This structure is derived from Element
| Name | Flags | Card. | Type | Description & Constraints |
|---|---|---|---|---|
 | 0..* | Element | Base for all elements | |
 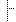 | 0..1 | date | ||
 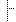 | 0..1 | date | ||
  | 1..1 | BackboneElement | ||
  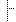 | 1..1 | string | ||
  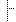 | 1..* | string | ||
   | 1..1 | code | ||
   | 1..1 | string | ||
   | 1..1 | date | ||
   | 1..1 | Element | ||
 Documentation for this format Documentation for this format | ||||
This structure is derived from Element
Differential View
This structure is derived from Element
Snapshot View
Other representations of profile: CSV, Excel
| Id | Grade | Path | Details | Requirements |
| ele-1 | error | CancerReport | All FHIR elements must have a @value or children : hasValue() or (children().count() > id.count()) | |
| ele-1 | error | CancerReport.extension | All FHIR elements must have a @value or children : hasValue() or (children().count() > id.count()) | |
| ext-1 | error | CancerReport.extension | Must have either extensions or value[x], not both : extension.exists() != value.exists() |